 京都大学の研究グループは,細胞にとって有害な構造異常糖タンパク質を小胞体から抹消する仕組みについて,従来のモデルを一新する提唱をした(ニュースリリース)。
京都大学の研究グループは,細胞にとって有害な構造異常糖タンパク質を小胞体から抹消する仕組みについて,従来のモデルを一新する提唱をした(ニュースリリース)。
分泌タンパク質や膜タンパク質が生合成される小胞体では,タンパク質の厳密な品質管理が行なわれており,正しい構造を獲得しないタンパク質は不良品として分解処理される。小胞体で合成されるタンパク質の多くには糖鎖が付加される。これら糖タンパク質が正常な構造を獲得できない場合,分解の目印として,糖鎖に含まれる9個のマンノースが7個以下へと刈り込みされる。
高等動物では糖鎖刈り込み酵素の候補分子として,EDEM1,EDEM2,EDEM3が注目されてきたが,実際に糖鎖刈り込み酵素活性を有するのか,それとも単純に糖鎖を認識する分子として機能するのか,10 年来議論されてきた。特に,EDEM2には糖鎖刈り込み酵素活性がないことが通説となっていた。
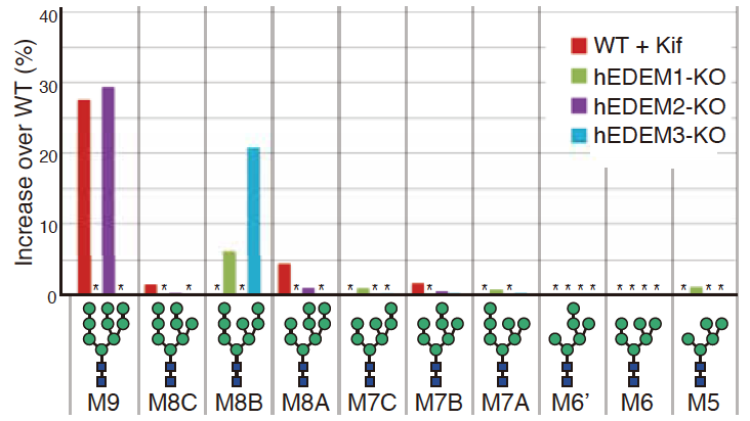
研究グループは,最近開発された革新的なゲノム編集法である Transcription activator-like effector nuclease(TALEN)法を活用し,これまで困難であったヒト培養細胞を用いた遺伝子破壊解析を行なった。その結果,EDEM1,EDEM2,EDEM3全てが糖鎖刈り込み活性を有することを初めて実証した。
また,各EDEMタンパク質の酵素活性には基質特異性があることを発見し,糖鎖刈り込みの第1段階(9個のマンノースを持つ糖鎖からマンノース1個を刈り込む反応)は,EDEM2により行なわれること,第2段階(8個のマンノースを持つ糖鎖からマンノースを1個以上刈り込む反応)は,EDEM1あるいはEDEM3により行なわれること,従来はEDEM1が最も重要な分子として捉えられていたが,EDEM1よりEDEM3の寄与が大きいことを明らかにした。さらに,EDEM2による糖鎖刈り込みの開始が,構造異常糖タンパク質分解経路に必須であることも明らかにした。
EDEMタンパク質の酵素活性の有無に関する長年の議論に決着をつけたこれらの成果は,これまでの糖鎖刈り込みモデルを一新した。今回の発見は小胞体における構造異常糖タンパク質分解機構を理解する上で重要なもの。
構造異常タンパク質の産生や分解処理機構の破綻はフォールディング病と総称される様々な疾患に関与することが知られており,研究グループではこの成果が,これら疾患の発症機構の解明や新しい治療戦略の立案にもつながると期待している。