 東京大学の研究グループは,古典的な生体内タンパク質のモデルにおいて,酵素反応を介在させることで,それぞれの反応が速くても,全体としての速度が数十万倍以上も遅くなりうることを理論的に初めて示した。(ニュースリリース)。
東京大学の研究グループは,古典的な生体内タンパク質のモデルにおいて,酵素反応を介在させることで,それぞれの反応が速くても,全体としての速度が数十万倍以上も遅くなりうることを理論的に初めて示した。(ニュースリリース)。
その多くが1秒以下で進む生化学反応を支配する時間スケールと,生物が生きている時間スケールの間には,大きな隔たりがある。多くの生化学反応が集まって構成されている生物が,どのようにその時間スケールのギャップを埋めているかは,ほぼ何もわかっていなかった。
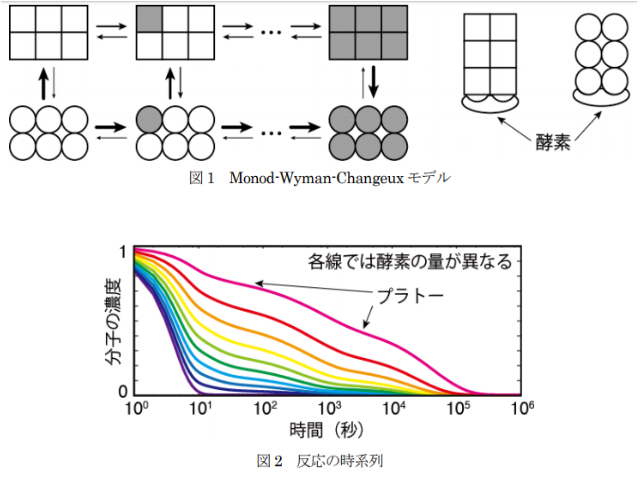
研究グループは,Monod-Wyman-Chnageuxモデルという生体内タンパク質のアロステリック制御のモデルを,実際に生体内のさまざまな反応で行なわれているように,酵素反応を含む形へと拡張し解析した。
その結果,時間スケールを遅くするメカニズムは,反応が進むほど,酵素と結合しやすい状態の分子が増えていく一方で,それらの分子が酵素を独占するような状態になってしまうために,残りの分子が酵素とほとんど結合できずに全体として反応が遅くなるというものだった。
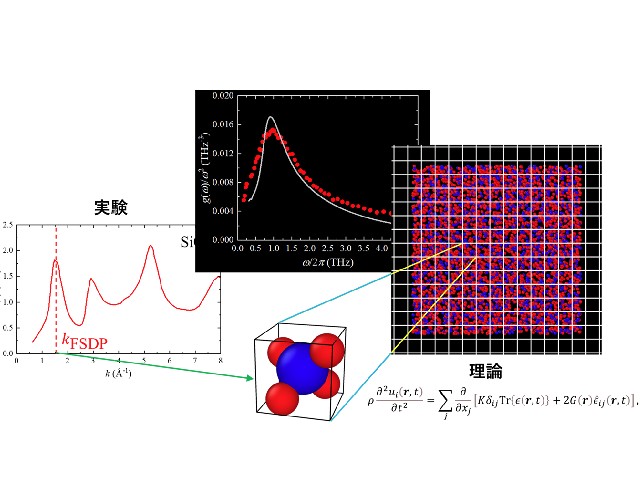
さらに,このゆっくりと進む生化学反応において,各分子の数がどのように変化していくかを調べたところ,各分子の数は,最終的に一定の割合に落ち着くまでに多数のプラトー(時間的にほとんど変化しない部分)を示しながら,時間に対して対数的に変化した。この時系列は,ガラス中の分子の運動に非常に近い。
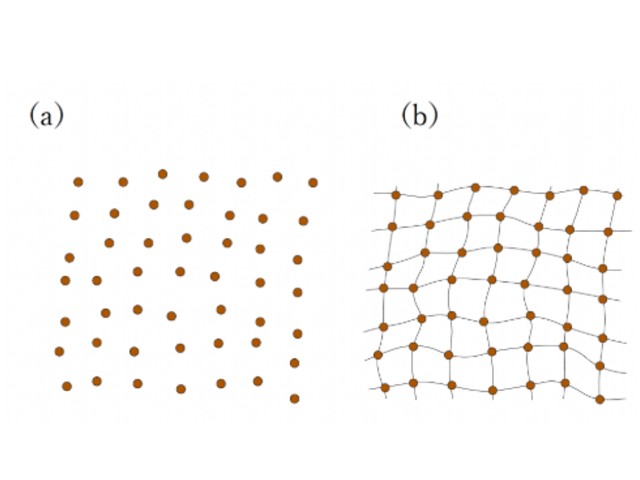
そこで,この生化学反応のモデルをさらに深く調べたところ,ガラスを支配している仕組みの一つと考えられる,動的拘束模型と非常によく似た構造が潜んでいることがわかったという。
動的拘束模型とは,エネルギー的に安定な状態はシンプルであるものの,そこに至るまでの状態の遷移できる状態に制限があるために,結果として安定な状態に至るまでの速度が遅くなるようなモデル。生化学反応においては,酵素が結合しないと反応速度がほぼゼロになるために,動的拘束模型のような仕組みが実現される。
また,ガラスでは環境が変化した時に,液体とガラスでの転移が起こる。そこで,酵素の量などを変化させたところ,反応が進んで最終的に行き着く状態は酵素の量に依存しないにも関わらず,その状態に至るまでにどのような反応経路を通るかは酵素の量に大きく依存した。
さらに,反応が全て終わるまでにかかる時間は,酵素の量と温度を変えた際に,それぞれ統計力学における一次転移と二次転移に近い振る舞いを示すことがわかった。この転移の存在は,反応のわずかな揺らぎにより各細胞に流れる時間が大きく変化し,細胞ごとの多種多様な応答を示しうることを意味するという。
時間スケールが重要な意味を持つ生命現象は,体内時計や睡眠,記憶など多岐に渡る。この研究は,これらの生命現象を支配する時間がどのように決まっているのかを理解するための,大きな礎となるとしている。