 国立遺伝学研究所の研究グループは,真核光合成生物の増殖機構の理解に向け,微細藻類の日周における細胞周期トランスクリプトーム変動解析を行なうと共に,遺伝子発現データベースを構築した(ニュースリリース)。
国立遺伝学研究所の研究グループは,真核光合成生物の増殖機構の理解に向け,微細藻類の日周における細胞周期トランスクリプトーム変動解析を行なうと共に,遺伝子発現データベースを構築した(ニュースリリース)。
水圏では様々な系統の微細藻類が光合成を行なっており,一次生産者として重要な役割を果たしている。近年,再生可能エネルギー,タンパク質源として藻類バイオマスの活用が注目されている一方で,水域の富栄養化によるプランクトンの異常発生(アオコの発生や赤潮など)など生活環境や水産業への被害も問題となっており,その増殖機構を知ることが重要となっている。
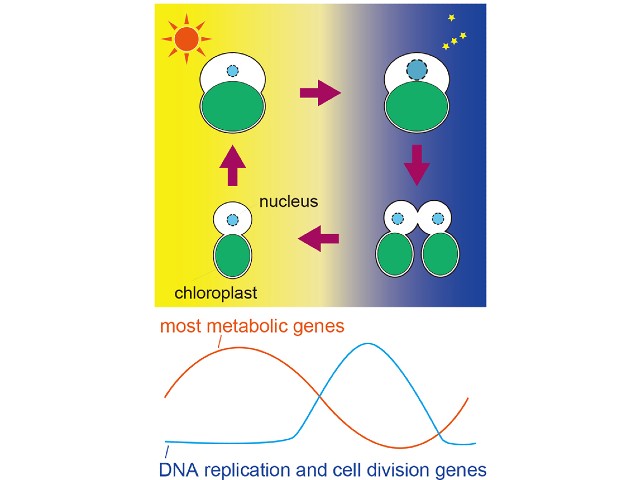
微細藻類は光合成によって増殖するが,自然環境には昼夜があり,光合成のできる時間帯は昼間に限られている。一方で,多くの微細藻類は夜間にDNA複製と細胞分裂(細胞周期進行)を行なうことが知られている。しかしながら,微細藻類が一日の中で,どのように効率よく光合成を行ない,どのような分子基盤で細胞分裂を行うなのか,という基本的な問題はほとんど理解が進んでいない。
この問題を解決するために研究では,微細藻類の日周におけるトランスクリプトーム変動解析を行なった。また,モデル藻類として単細胞紅藻Cyanidioschyzon merolae(シゾン)を用いた。この藻は遺伝子数が真核光合成生物の中で最少クラス(16Mb,約5,000遺伝子)で,解析対象となる遺伝子セットが極めて少なく様々な実験に有用。
解析の結果,昼間には多くの主要代謝系(炭化水素,アミノ酸,ビタミン合成など)の遺伝子群が一斉に誘導されること,一方で夜間には解糖系に関連する一部の遺伝子群やdNTP(DNAの材料)新生に関わる遺伝子群が誘導されることがわかった。この結果は昼間に光合成を行ない細胞生長し,夜間に細胞周期進行することをよく反映している。
また,細胞周期に依存する遺伝子発現を日周期によるそれと区別するために,細胞周期制御タンパク質の変異体を作成し,トランスクリプトーム変動解析を行なった。その結果,既知の細胞分裂関連遺伝子群とともに,細胞周期に依存する機能未知遺伝子群を見つけることが出来た。これらは,藻類の細胞分裂に関わる新規遺伝子群である可能性が高いと考えられるという。
今回の研究で樹立した遺伝子発現データベースは,微細藻類の日周における基本的な細胞動態の理解にとどまらず,将来的には,代謝改変によるバイオマス生産の増大,プランクトンの異常発生の予測や対策などに役立てることが出来るとしている。