京都府立医科大学の研究グループは,蛍光顕微鏡画像用にデザインされた「ボリュームレンダリングソフトウェア」を開発した(ニュースリリース)。
蛍光顕微鏡や組織透明化技術の進歩に伴い,蛍光情報を保ち組織・臓器・胚の全体を顕微鏡による観察が可能となる一方,顕微鏡から得られた断層像を用い,蛍光染色による断層画像から三次元的観察や解析を行なうには,非常に高額な,高性能なワークステーションや画像処理用演算プロセッサ(GPU)と専用のシステムが必要だった。
研究グループは,米AppleのMacに一般的に装備されているApple Siliconが有するユニファイドメモリの設計の特性を活用した,新規のボリュームレンダリングソフトウェア「Acto3D」を設計し開発した。
ユニファイドメモリでは,従来の専有GPUと異なり,GPUからシステムメモリ領域へ直接的かつ高速にアクセス可能であることから,大容量の専有メモリを備えたGPUであると捉えることができる。
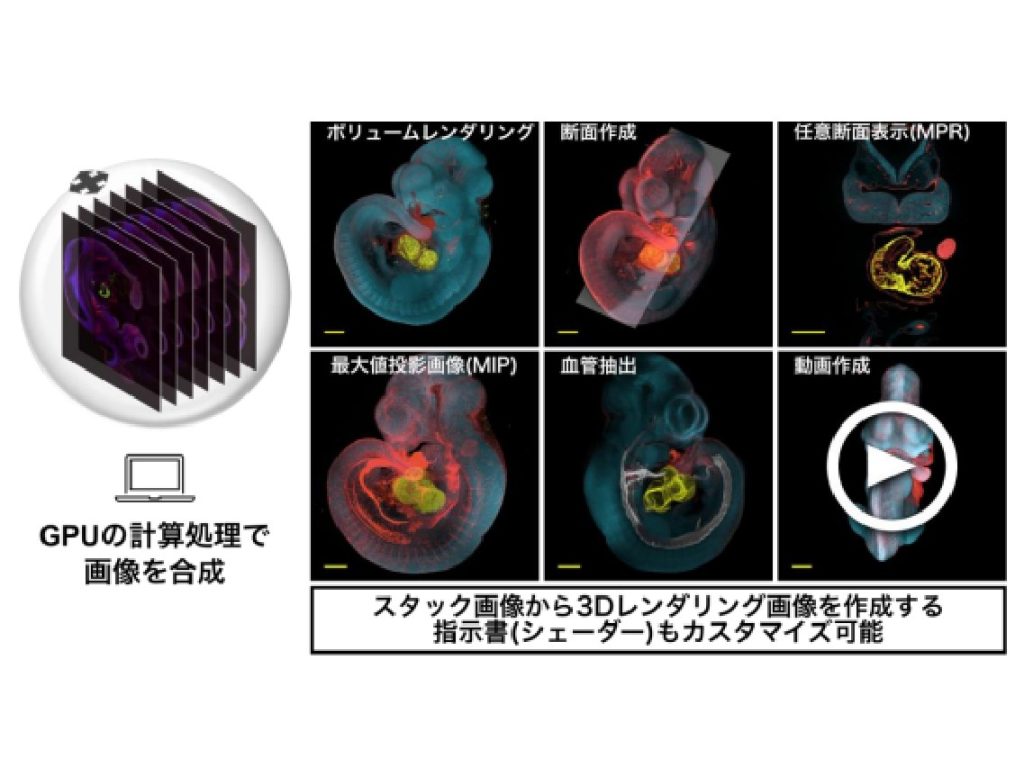
この特性を利用し,蛍光顕微鏡での撮像データ全体をメモリ上へ展開し,ピクセル値を高い自由度で高速に合成することで,MacBook Proなどのラップトップマシンでも高解像な顕微鏡画像の三次元観察を可能とした。Macに特別な環境構築を行なう必要がなく,撮像したデータセットを容易に空間上で任意の視点から観察できるという。
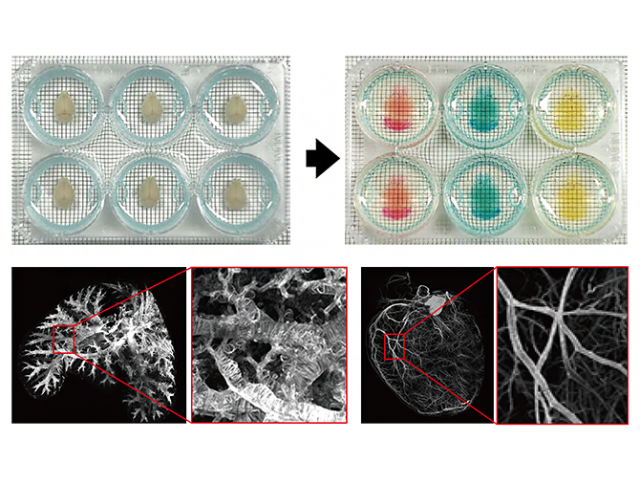
実際のマウスの受精後12.5日目の胚の心臓,心筋タンパクに対する抗体,刺激伝導路を構成するHCN4に対する抗体と核を染色し,CUBIC法と呼ばれる組織透明化法を用いて心臓そのものを透明化し,光シート顕微鏡で画像を取得した。
これらのデータをActo3Dで立体画像構築したところ,従来のソフトと比較し,①高精細,②自由な観察断面が得られる,③特定のタンパク染色像を抽出できる,④切片作成が不要で組織の非可逆的な損傷が避けられる,などの利点を得確認した。
さらに三次元画像解析の例として,教師なし機械学習の手法を応用して,空間画像データセット上での管腔構造の抽出アルゴリズムを提唱し,複雑なマウスの大血管の立体構造を抽出することにも成功した。
このように構築した咽頭弓動脈のモデルは従来の連続切片を手動でトレースする方法に比べると,正確かつ高精細であり,他の抗体染色と組み合わせることで周辺の組織構造と関連付けて観察することが可能だという。
Acto3DはmacOS限定だが,手持ちのMシリーズのMacで動作するため,新規の投資を最小限に抑えつつ,多くの研究者に空間観察・解析のリソースを提供するとしている。