東邦大学と名古屋工業大学は,紫外線損傷DNAを光で修復する酵素活性をもつDNA配列(DNA酵素)を対象として,ナトリウムイオン濃度依存的な構造変化を見出し,また,損傷DNAの光修復における構造変化を計測することに成功した(ニュースリリース)。
生体内で遺伝物質として機能しているDNAは,化学物質や活性酸素に曝されたり紫外線を照射されたりすると損傷を受ける。DNA損傷のうち,隣り合ったピリミジン塩基間での共有結合の形成が紫外線によって引き起こされる。
生物が備えているDNA修復機構の1つにDNA光回復酵素によるものがある。DNA光回復酵素は,近紫外線や青色の光を使って塩基間の共有結合を切断して元の正常な塩基に戻す反応を触媒する。
この研究では,損傷DNAを近紫外線や青色の光を用いて修復するDNA光回復酵素と同じ働きをもつDNA配列を対象とした。酵素活性をもつDNA配列なのではDNA酵素と呼ぶ。
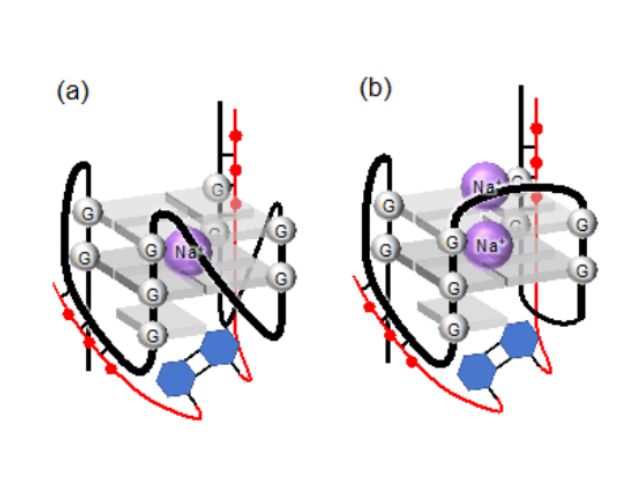
UV1Cと名付けられたこのDNA酵素はナトリウムイオン存在下で平行グアニン四重鎖と呼ばれる特殊な構造を形成し,通常の構造のDNAが吸収しない305nmの光を吸収して損傷DNAを修復することが報告されている。
研究グループは,過去にタンパク質酵素であるDNA光回復酵素の研究を行なっており,その修復機構をDNA光回復酵素の解析に用いた手法と同じフーリエ変換赤外(FTIR)分光法を用いて解明することを試みた。
はじめに,UV1Cによる損傷DNAの修復が正しく再現されるかの確認と条件検討を行ない,確かにUV1Cはナトリウムイオン存在下で損傷DNAを光依存的に修復することを示した。
次いで,ナトリウムイオン濃度を変えたときにグアニン四重鎖がどの程度形成されるかを円偏光二色性を用いて計測した。それまで240mMという特定のナトリウムイオン濃度だけで活性を観測していたのに対し,研究グループはFTIR分光法で計測することを念頭に,より適切なナトリウムイオン濃度がないかを調べた。
その結果をもとに,ナトリウムイオン濃度を240mMと1500mMにした状態で損傷DNAの修復に伴う構造変化をFTIR分光法で観測した結果,ハイブリッド型グアニン四重鎖のほうが平行グアニン四重鎖より構造的なひずみが大きいことを示唆する結果を得た。
今回研究グループは,DNA酵素 UV1Cの構造の一端を解明した。DNAは一般に乾燥や熱に対して高い安定性をもっており,将来的にはこのDNA酵素を改良することで,生体内での紫外線損傷DNAを修復するサンスクリーン剤などへ応用できるとしている。