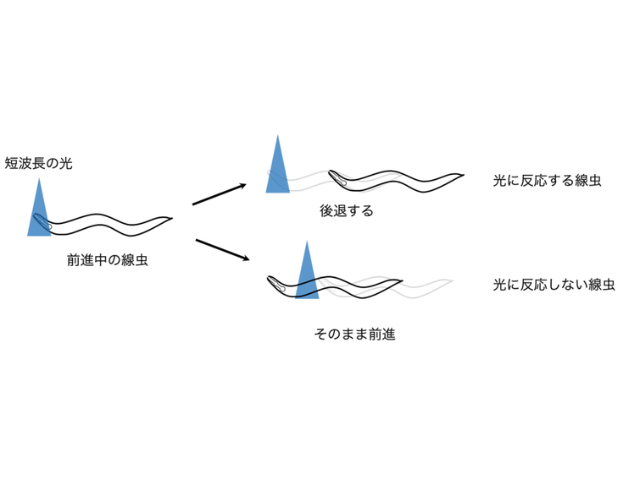
 東京大学の研究グループは,線虫の神経細胞の配置を調べて,個体ごとに配置が大きくばらつくことを見出し,配置がばらついていても各々の神経細胞を同定できる方法を開発した(ニュースリリース)。
東京大学の研究グループは,線虫の神経細胞の配置を調べて,個体ごとに配置が大きくばらつくことを見出し,配置がばらついていても各々の神経細胞を同定できる方法を開発した(ニュースリリース)。
ある遺伝子の塩基配列に蛍光タンパク質をつなげて,発現が見られた神経細胞の配置や形状をアトラスと比較することで,その遺伝子を発現する細胞を同定できる。このように特定の細胞で発現を誘導する塩基配列は細胞特異的プロモーターと呼ばれ,蛍光タンパク質の代わりに遺伝子コード型カルシウムセンサーをつなげば,発現細胞の神経活動を測定することもできる。
一方,近年の顕微鏡技術の発展により,一個体の脳全体を一細胞レベルで可視化する全脳イメージングが可能になった。線虫は体長約1mmと小さく,体が透明なので,頭部の全神経活動を一細胞レベルで観察することができる。
しかし全ての神経細胞が光ってしまうと個々の細胞の形状が得られないなど,全脳イメージングを行った個体内の個々の神経細胞を同定する手法がなく,個体間で神経活動を直接比較することは難しかった。
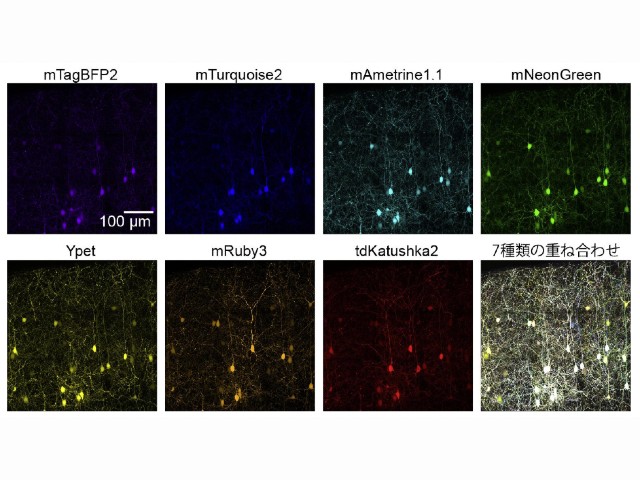
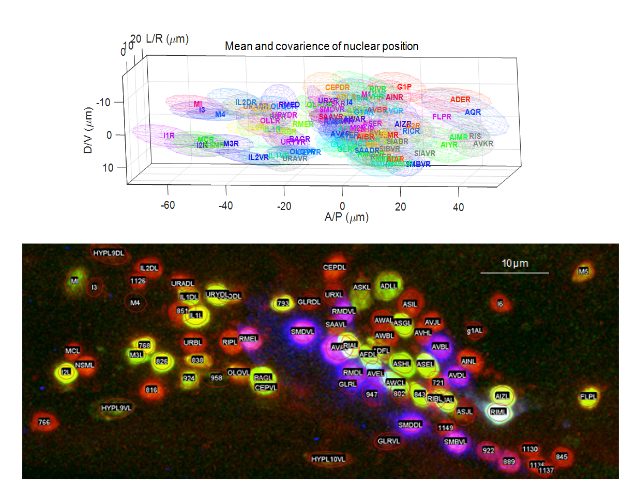
今回,研究グループは,特定のパターンで蛍光タンパク質を発現する35種類の線虫株を作出し,311匹の線虫の頭部を詳細に観察した。蛍光タンパク質を発現した細胞をランドマークとして,頭部のほぼ全ての神経細胞を網羅的に同定した。隣接した細胞の距離は平均3.2μmだったが,各細胞の位置は平均2.7μmほどばらついていることを見出した。
こうした個体間のばらつきのため,細胞の配置だけを頼りに細胞を同定することは難しいことがわかった。そこで発現パターンの組み合わせを探索して,細胞同定に最適な線虫株を作出した。
この線虫株ではひとつの個体内で同定できる細胞の数が従来の3.6倍に増え,線虫頭部のほとんどすべての神経細胞に名前がつけられるようになった。さらに全神経活動の観察ができる線虫株とかけあわせた株でも同様に細胞が同定できることを確かめ,全脳神経活動の同時観測と細胞同定とを統合することに成功した。
また研究グループは,神経細胞を自動的に同定するソフトウェアを作成した。観察者による修正をフィードバックすることで,正しい細胞同定結果を半自動的に得ることもできた。
今回の研究によって,全神経活動を同時に計測した結果を神経回路に対応付けて,個体間で比較するための基盤的技術が得られた。この研究の成果は,包括的な観察を通じた神経回路の動作原理の解明などに役立つとしている。