 東京理科大学の研究グループは,巨大ウイルスに人為的に感染させた単細胞生物アカントアメーバを,位相差顕微鏡を用いて経時的に撮影し,得られた画像を解析して,細胞の大きさや形状の変化,数や動きなどを定量化できるプログラムを開発した(ニュースリリース)。
東京理科大学の研究グループは,巨大ウイルスに人為的に感染させた単細胞生物アカントアメーバを,位相差顕微鏡を用いて経時的に撮影し,得られた画像を解析して,細胞の大きさや形状の変化,数や動きなどを定量化できるプログラムを開発した(ニュースリリース)。
位相差顕微鏡では,たとえ試料が透明であっても明暗のコントラストを持った像が得られる。細胞を染色することなく,自然な動態を観察することができるため,細胞生物学や臨床医学などの分野で特に有用となる。
しかし,光の位相差を利用する特性のため,試料の輪郭にオーラ状に光が染み出して見えるハローや,大きな試料の内部の明るさが背景と同化してしまうshade-offが起こりやすく,このことが解像度の低下や,既存の画像解析技術の適用の難しさにつながっていた。
研究グループは,巨大ウイルスの自然環境中の分布と,感染が生物の生存や進化に与える影響について研究している。今回,アカントアメーバに巨大ウイルスをそれぞれ感染させ,位相差顕微鏡を用いて撮影を行なった。
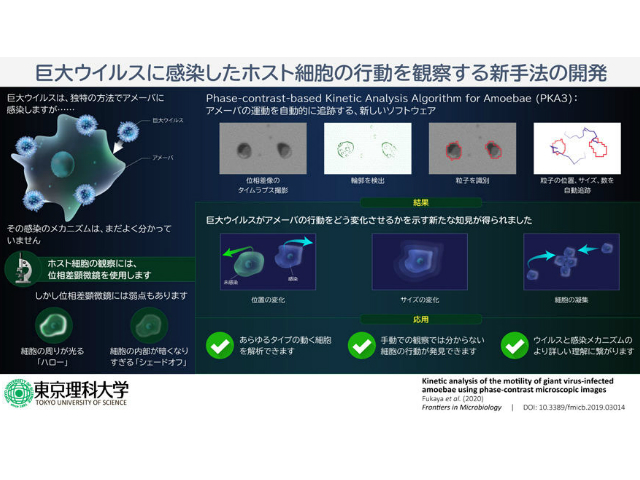
得られた画像から細胞を識別するために,画像上で強度が不連続に変化する場所を試料の輪郭として捉え,何重もの複雑な輪郭に囲まれた領域の中からハローらしい部分を除去し,粒子らしい部分のみを検出した。しかしこの検出方法は,ハローを無視できるわけではないため,粒子の細かい形を高い解像度で観察することには適さない。
しかし,粒子の数を数え,粒子の大きさや円形度を算出することは十分可能で,さらに,各画像を次の画像と比較して最も近い位置にある粒子同士を,同一の細胞が移動したものとみなすことで,細胞が移動した距離を定量的に測定することもできるようになった。
このタイムラプス撮影された複数の位相差顕微鏡画像を使い,C言語およびC++言語を使用したプログラム「位相差を利用したアメーバの動態観察アルゴリズム(PKA3:Phase-contrast-based Kinetic Analysis Algorithm for Amoebae)」により,アカントアメーバと,アカントアメーバに感染した巨大ウイルスのサイズや形状,数,動きなどを定量化することに成功した。
今回のPKA3に用いられた技術は,位相差顕微鏡で観察可能なすべての細胞に応用できるため,今後の細胞生物学の発展に大きく貢献するとしている。