 新潟大学の研究グループは,細胞の3D構造をライブで可視化する新規の顕微鏡観察法を開発し,細胞の形と動きを自動解析する技術を確立し,これを用いて神経細胞および細胞性粘菌が,キラルな旋回・らせん運動を示すことを発見した(ニュースリリース)。
新潟大学の研究グループは,細胞の3D構造をライブで可視化する新規の顕微鏡観察法を開発し,細胞の形と動きを自動解析する技術を確立し,これを用いて神経細胞および細胞性粘菌が,キラルな旋回・らせん運動を示すことを発見した(ニュースリリース)。
人体の器官の多くはほぼ左右対称な配置をとっているが,内臓の一部や脳において,機能的な左右差があることが知られている。しかし,脳の左右差が生じる理由は分かっていないが,細胞のキラリティが左右非対称性の形成メカニズムを解き明かす鍵として注目を集めている。
キラリティと非対称性形成のメカニズムを解明するためには,キラル現象の正確な情報が必要だが,1)細胞を3Dライブイメージングにより精密に計測し可視化する技術,2)高次元の画像データから形と動きを自動解析する技術,はこれまで存在しなかった。
細胞のライブイメージングには蛍光顕微鏡を使用するのが一般的だが,高頻度3D撮影すると,通常の生細胞では,励起光に起因する光毒性により構造が簡単に壊れてしまう。一方,微分干渉顕微鏡は,無染色・低毒性で高解像度撮影が可能だが,陰影の付いた画像しか得られず,そのままでは3D可視化・解析に使えない。
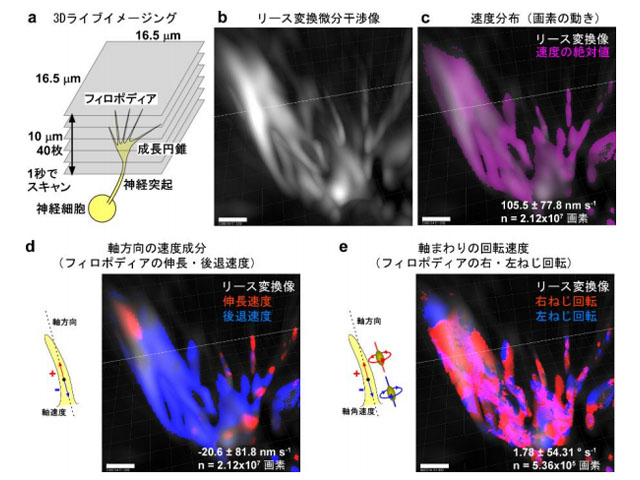
研究グループは,リース変換と呼ばれる特殊な数学的操作を施すことで,微分干渉像に含まれる陰影付きの位相情報を輝度情報に変換する方法を考案し,「リース変換微分干渉顕微鏡法(RT-DIC)」と名付けた。この手法を使うことで,蛍光像とほぼ等価な,明るく光る輝度画像を得て,脆弱な細胞をライブで3D可視化することに成功した。
さらに,多次元画像データから細胞の形と動きを自動解析する手法の開発を行なった。コンピュータービジョンの技術を取り入れ,画素ごとの形状と運動の物理量を数値的に推定する手法を確立した。さらに,画素ごとの形状情報と運動情報を時空間的に統合することにより,粒子・細胞・線維の走行経路・運動軌跡を追跡することに成功した。
開発した技術を,伸びていく神経の先端構造で伸びる方向の制御に重要な,神経成長円錐のキラリティ解析に応用した結果,神経成長円錐の3D形態と動きをとらえることに世界で初めて成功した。それらの形と動きを数値化することにも成功し,神経成長円錐の運動様式を明らかにすることができた。
さらに,粘菌細胞はアメーバ状の単細胞期において,2D基質上では細胞が右回り(時計回り)に旋回しながら移動,3D環境だとフィロポディア様の突起が右ねじ回転していることがわかった。
今回の技術により,これまで扱いの難しかったキラリティをシステマティックな手法で定量的に解析することが可能になり,細胞のキラル現象を正確に記述し特性を明らかにできる。この成果を応用して,左右非対称性形成のしくみが解明されることが期待されるとしている。