理化学研究所は、薬剤の作用を遺伝子発現量の変化(プロモーター活性の変化)として網羅的・定量的に捉えることに初めて成功した。
理研が開発した独自技法「非増幅deepCAGE法」は、細胞中のmRNAを少量でも偏りなく網羅的に捉え、遺伝子の転写開始点の直上流にあるプロモーター活性を測定できる。さらに、塩基配列を高速に解析できる次世代シーケンサーを用いることで、解析対象の細胞に存在する全mRNAの種類と量が解析可能となる。
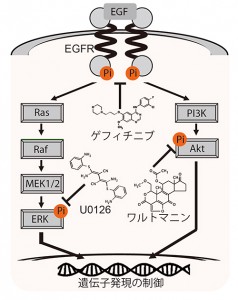
共同研究グループは、非増幅deepCAGE法を用いて、抗がん剤を投与する前と投与した後のがん細胞のプロモーター活性を解析した。その結果、薬剤の作用により個々のプロモーター活性が促進あるいは抑制されたかを、定量的かつ感度良く捉え、2次元上のグラフ(プロモーター活性プロファイル)として示すことができた。
また、標的分子が異なる2種類の抗がん剤によるプロモーター活性変動の相違は、2つのプロファイルの比較で明瞭に観察できた。さらに、2種類の抗がん剤のプロモーター活性プロファイルを組み合わることで、同じ情報伝達経路内の別の分子に作用するもう1種類の抗がん剤のプロモーター活性プロファイルを表せることも分かった。これは、既知の薬剤のプロモーター活性プロファイルから、未知の薬剤の作用を推定することが可能であることを示している。
これらの結果は、非増幅deepCAGE法を用いた遺伝子発現の定量解析が、既存の薬剤の標的タンパク質や作用機序の解明などに広く応用可能であり、新薬開発での薬理学分野への貢献が期待できることを示している。
詳しくはこちら。