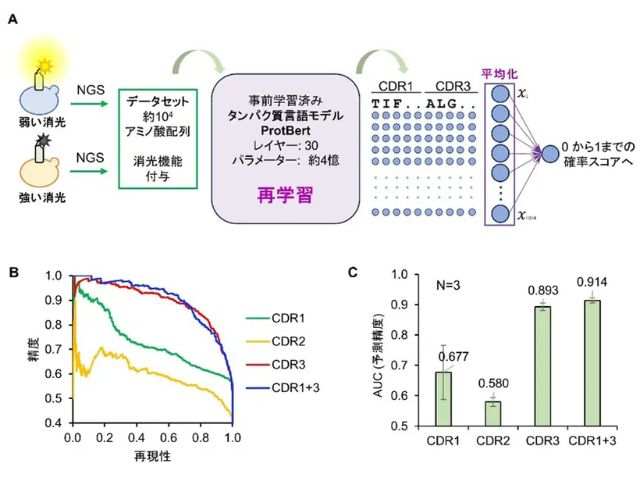
 理化学研究所(理研),東京大学,京都大学は,新型コロナウイルス(SARS-CoV-2)由来のウイルスRNAを「1分子」レベルで識別して5分以内に検出する技術を開発した(ニュースリリース)。
理化学研究所(理研),東京大学,京都大学は,新型コロナウイルス(SARS-CoV-2)由来のウイルスRNAを「1分子」レベルで識別して5分以内に検出する技術を開発した(ニュースリリース)。
今回,研究グループが開発した技術は,理学と工学の異分野融合研究によるもので,その手法を「CRISPR-based amplification-free digital RNA detection;SATORI)法」と名付けた。
SATORI法は,「マイクロチップを利用した酵素反応の1分子検出技術」と「核酸切断酵素CRISPR-Cas13a(Cas13a)」に関する技術を融合させたものであり,特定のRNA配列を認識するCas13aと蛍光レポーターの混合液をバイオセンサーとして利用することで,検体中の標的ウイルスRNAの有無を高感度・高精度・迅速に検出する。
SATORI法によるウイルスRNAの検出原理は以下の通り。
① 核酸切断酵素Cas13aと蛍光レポーターの混合液にウイルスRNAを混ぜると,特異的にウイルスRNAとCas13aの複合体が形成される。
② 複合体が形成されるとCas13aの酵素活性がオンとなり,蛍光基と消光基がつながった蛍光レポーターが切断される。
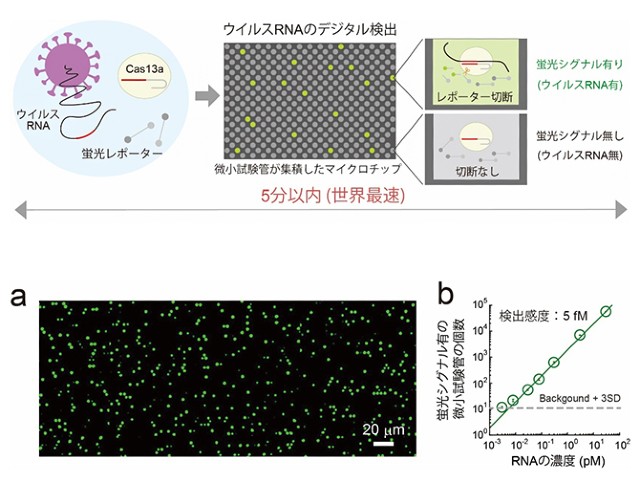
③ ②の複合体と蛍光レポーターの混合液を,3fLの微小試験管が100万個集積されたマイクロチップアレーに小分けにして封入すると,Cas13aの切断活性に伴いウイルスRNAが存在する試験管だけ蛍光シグナルが1分以内に大きく上昇する。
④ 蛍光シグナルの有無を二値化し,そのデジタル信号からシグナル有の微小試験管の個数をカウントする。カウントされる試験管の個数が検体中のウイルスRNAの個数に相当するため,ウイルスRNAの存在を1分子レベルで判別・検出できる。
これにより5分以内にウイルスRNAを1個ずつ識別して検出できる。検出感度は5fMであり,これはウイルスRNAの量で表すと,1μLあたり約103個となる。この感度は従来の抗原検査法の104~105個/μLと比較すると10~100倍高く,PCR検査の10~102個/μLと比較すると10~100倍低いということになる。
ただし,SARS-CoV-2感染者の検体中のウイルスRNA量は,103~106個/μLであることから,SATORI法はSARS-CoV-2の感染診断を実施する上で必要な感度を満たしているとする。
ランニングコストはおよそ9ドルでPCR検査の5ドル程度とほぼ同等であるため,今後,消耗品の大量生産や検出装置の小型化により,安価で素早く多種のウイルス感染症を正確に診断できることが期待できるとしている。