 東邦大学は,新しく開発した短い配列のDNAを用いて高感度な生体分子を検出する方法(in situ hybridization chain reaction法,以下in situ HCR法)を開発した(ニュースリリース)。
東邦大学は,新しく開発した短い配列のDNAを用いて高感度な生体分子を検出する方法(in situ hybridization chain reaction法,以下in situ HCR法)を開発した(ニュースリリース)。
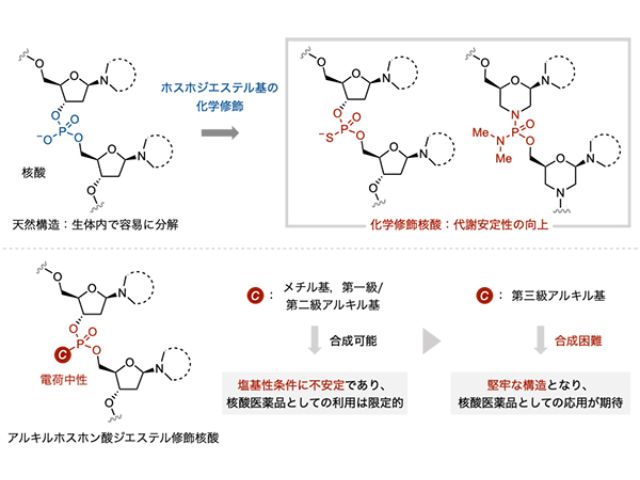
組織における様々な遺伝子の発現やタンパク質量のモニターは生命現象を理解する上では無くてはならない。mRNAはタンパク質などの物質合成に関わり,細胞の役割を決定付ける。これまでのmRNAを検出する方法は検出感度が低く,時間がかかるなどの欠点があった。また,近年開発された,人工合成したDNAを用いてmRNAを高感度に検出する手法の多くは非常に高額で,普及の壁となってきた。
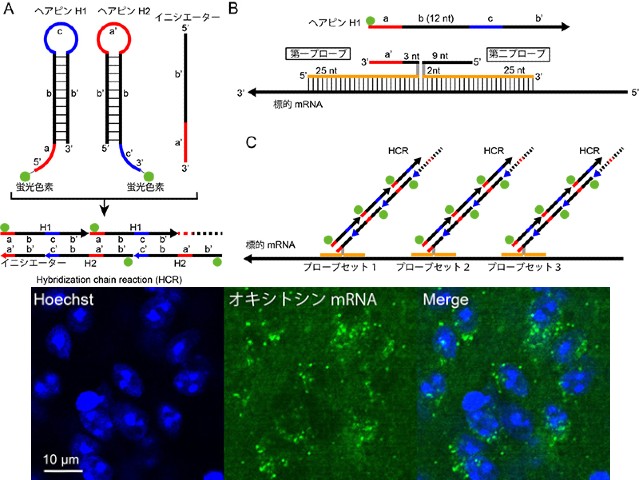
in situ HCR法は蛍光標識されたヘアピン型の合成DNAを用いる手法として知られている。この手法は、組織内でヘアピンDNAを非酵素的に重合させることで1分子のmRNAを検出できる感度を持ち,検出までにかかる時間が短いという利点を持るが,検出に用いるヘアピンDNAの鎖長が長く,純度の高いDNAの合成が難しいため高額なものだった。
そこで研究グループは,このヘアピンDNAの鎖長を短くする試みを行なった。DNAの鎖長を減らすとDNAのヘアピン構造の安定性が低下し非特異的な反応が起こることや,重合反応の効率が落ちることが知られている。この問題を解決するため,短いヘアピンDNAであっても安定性を維持し重合反応の効率の高いDNA配列を複数見出し,その配列の法則を発見した。
また,DNAの標識に用いるアミノリンカーや蛍光色素についても最適な条件を見出すことに成功した。これらの改良により,DNAの合成にかかる費用は60%以上減少した。
この短鎖ヘアピンDNAを用いてmRNAの検出を試みたところ,既存の長いヘアピンDNAを用いた場合と同様に1分子のmRNA検出が可能で,さらに酵素を用いたmRNA検出よりも高感度であることが示された。また,同時に複数種類のmRNAを異なる波長の色素で検出することが可能で,これまで数日から1週間かかっていた検出操作が24時間以内に終わることも明らかになった。
これらの特長に加え,ヘアピンDNAの鎖長を短くしたことによりDNA分子の組織内への浸透性が向上し,細胞膜を破壊するような処理などを用いずともmRNAの検出感度が落ちないことも明らかになり,今後様々な研究に応用される基盤技術となることが期待されるとしている。