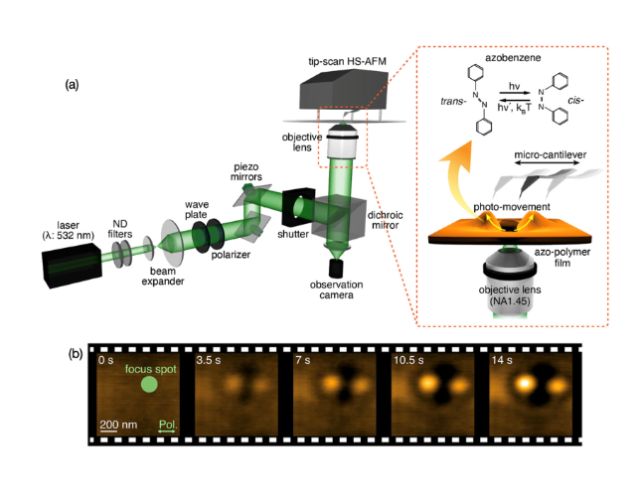
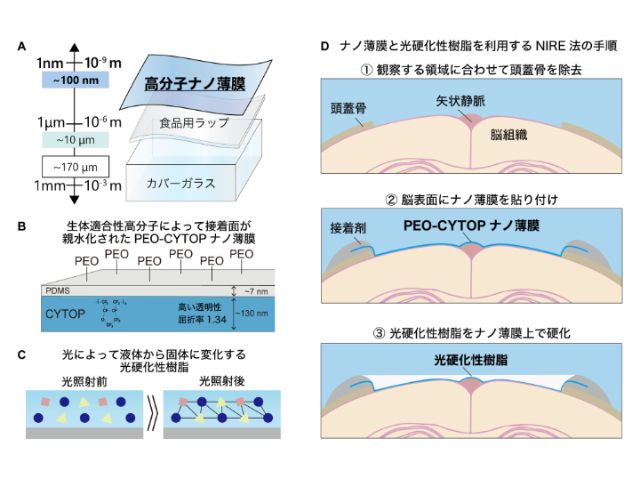
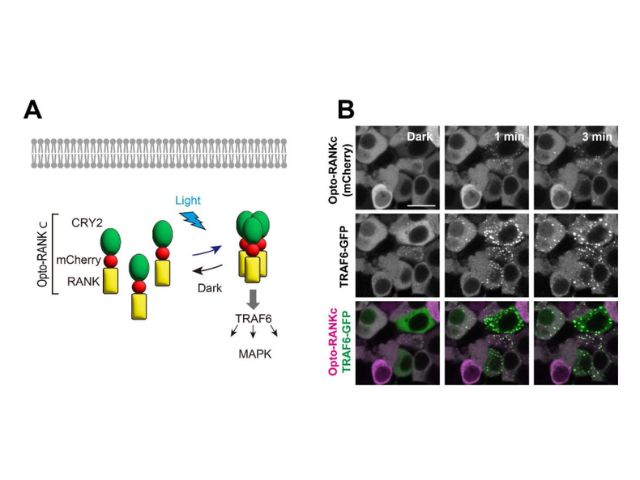
 東京大学の研究グループは,マウス全脳血管ネットワークの構造と機能を可視化する手法を開発した(ニュースリリース)。
東京大学の研究グループは,マウス全脳血管ネットワークの構造と機能を可視化する手法を開発した(ニュースリリース)。
脳は3次元上に広がる血管ネットワークによって栄養供給を受けている。また,血管と組織中の物質交換は,多様な細胞からなる血液脳関門によって制御されている。
そのため,血管ネットワークの構造と機能,それらの周囲組織との関係を記述することは,脳の栄養供給様式の理解に重要であることが推察されるという。しかし,このような研究は技術的な問題からこれまで進んでこなかった。
例えば,薄層切片を用いた古典的な組織学では血管は切断されるため,その接続関係の情報は失われてしまう。接続関係を調べるためには標本を3Dで観察する必要があるが,血管の3D観察に古くから用いられている血管腐食鋳造法は,微小血管の可視化に優れないのに加え,周囲の分子情報を保存できない,という問題を抱えている。
その点,組織透明化法は,標本を3D観察できるのみならず,血管周囲の分子情報を保存できるので,前述の問題を解決することが期待される。ただし,透明化手法は既存の血管染色手法とは相性が悪く,周囲分子を保存したまま全脳血管の構造を捉えることは困難だった。
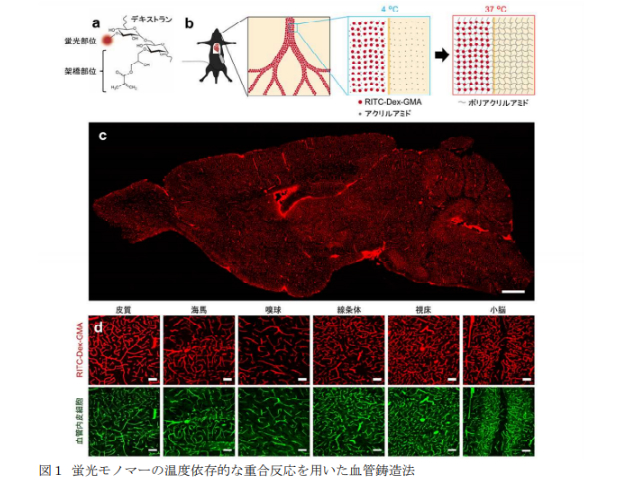
研究グループは,今回,独自の蛍光モノマーを用いた血管鋳造法と,それに最適化された組織透明化手法を開発した。この手法SeeNetは,ほぼ全ての脳血管を明瞭に可視化しつつ,周囲の組織中に存在するタンパク質の抗原性や,緑色蛍光タンパク質(GFP)等がもつ蛍光を保存していた。
更に,この手法の高いSN比で全脳血管を捉えることが可能か検討するために,ライトシート顕微鏡を用いて脳の撮影を行なった。結果,脳深部の白質においても血管のシグナルが捉えられており,脳の最も深い部分においてもシグナルが消失することはなかった。
このことから,この手法は全脳の血管を捉えることが可能であることが示され,応用例として,動脈と静脈を区別した全脳血管のイメージングにより,これまで未知だった,皮質と海馬の細動脈/細静脈を繋ぐ微小血管を報告した。
SeeNetは,発現分子が紐づけられた脳血管の3次元トレーシングや,血管の構造・機能と周囲の細胞との関係を調べるのに有用なツールとなり,脳梗塞や脳出血などの脳血管障害の病態解明に貢献するのみならず,機能的MRI信号の解読への一助になることが期待されるとしている。