沖縄科学技術大学院大学(OIST)は,細胞の遊走や形態の経時変化を定量化を可能にするツールを開発した(ニュースリリース)。
人体は多くの細胞で構成されている。一般的には,顕微鏡下で細胞を観察するためには,染色液で染めるか,遺伝子を操作して,鮮やかな色の蛍光ラベルを使用して発光させる必要がある。しかし,染色すると細胞の動きが変わるため,実験結果も歪められてしまう。
一方で,いわゆる「ラベルフリー」という手法を用いて,蛍光染色せずに細胞遊走の観測を試みる方法もあるが,ラベルフリーで標識のない細胞は,顕微鏡画像の背景に溶け込んでしまい,既存のソフトウェアで解析することは非常に困難だった。
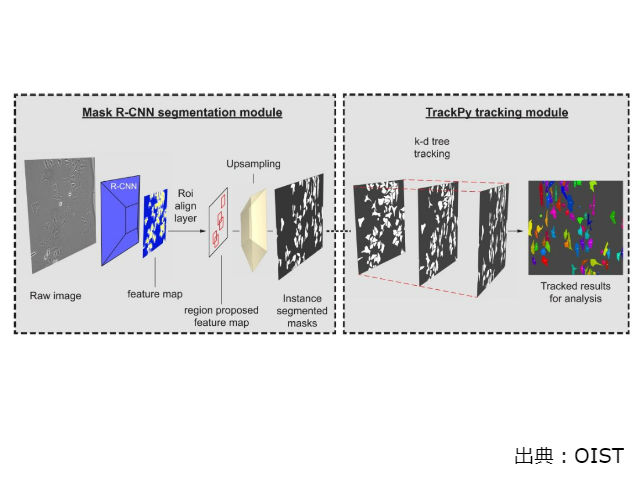
しかし,今回研究グループが開発した「ウシーガチ」を使ってソフトウェアに学習させると,研究者が教師役になり,ソフトウェアに新たな画像を学習させ,細胞を区別させることができるという。「ウシーガチ」は琉球の方言で,物体の輪郭をトレースする「透き写し」を意味する。
このプログラムは,脳細胞の情報処理方法に類似した「畳み込みニューラルネットワーク」という機械学習の仕組みを用いている。人間の目は光を捉えると,ニューロンがそのシグナルを分析し,目に映る物体とその位置関係を認識する。
ニューロンはまず,物体を大まかに描写し,情報を次のニューロンに伝え,この伝達を繰り返すうちに,画像はより詳細に描写されていく。ニューラルネットワークも同じ仕組みだが,唯一の違いは,ここでの「ニューロン」は実際の細胞ではなく,コードの集合体となる。
また,このソフトウェアは,ラベルフリーの細胞の遊走を,簡単にセグメント化,追跡,解析することができる。細胞迷走を定量化するための複数の機能を1つにまとめたソフトとして,もしくは,3つの個別のアプリ(セグメンテーション,トラッキング,データ解析)として使用可能。
研究グループは,今後,細胞の輪郭だけではなく,細胞内の構成要素を特定できるニューラルネットワークの開発を目指す。またこのツールは,顕微鏡で様々な細胞が,正常か異常か,新しいか古いかを容易に判定することや,細胞系譜まで分析できる可能性があるとしている。
なお,このソフトウェアの使用方法は動画説明付きでオンライン上に一般公開されており,誰でも利用することができる。