 名古屋大学および産業技術総合研究所は,100万原子以上を持つ分子を高速で解析する新しいコンピューターシミュレーション法を開発した(ニュースリリース)。
名古屋大学および産業技術総合研究所は,100万原子以上を持つ分子を高速で解析する新しいコンピューターシミュレーション法を開発した(ニュースリリース)。
生化学やナノエレクトロニクスの分野で扱う分子には,タンパク質やフラーレン群といった1万〜100万個という多数の原子からなるものが多く見られる。これまでの量子化学を用いてこれらの分子群を解析しようとすると,膨大なコンピュータメモリを消費し,長時間を要していた。そのため,原子量が多い分子の解析は非常に困難とされていた。例えは,、分子システムのサイズを100倍大きくした場合,消費するコンピュータメモリは100万〜100兆倍かかることもあった。
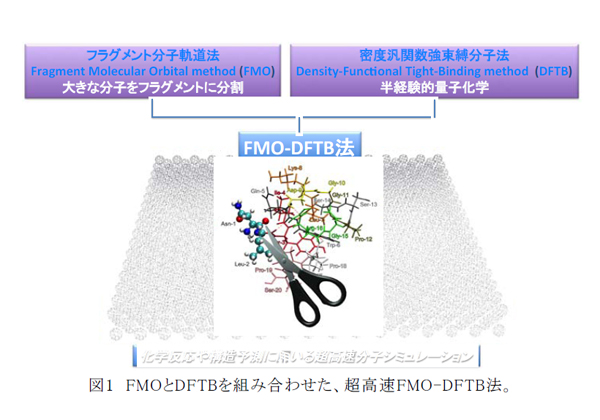
今回,研究グループは,大きな分子をフラグメントに分割するフラグメント分子軌道法(FMO)に密度汎関数強束縛分子法(DFTB)を組み合わせたFMO-DFTBという新たなコンピュータシミュレーション法を開発した。このFMO-DFTB法によって,DNA,タンパク質といった大型分子や100万原子を超えるフラーレン群の構造解析に成功した。また,約2万個の原子を有する水分子群をFMO-DFTB法を用いて解析したところ,DFTB法のみではおよそ3週間かかると予想される結果に対して,約3分間(これまでの約1万倍の速度)で解析結果を得ることができた。
FMO-DFTB法は,これまでスーパーコンピュータを必要とした巨大複雑系分子をデスクトップパソコンなどで高速,かつ精密に解析することができる。この新しい方法は,化学反応を始め,生体分子やナノエレクトロニクス材料の構造解析など,生物や化学をはじめとした様々な研究分野での応用が期待されるとしている。
なお,FMO-DFTB法は,GAMESS-US(無料量子化学ソフトウェア)の次回更新で提供される予定。
GAMESSホームページ: http://www.msg.ameslab.gov/gamess/
関連記事「NIMSら,従来比2桁以上の原子数を扱える第一原理シミュレーションを開発」「理研、海藻類の有機・無機成分複雑系の統合解析技術を構築」