 国立遺伝学研究所(NIG)は,蛍光顕微鏡画像を簡便かつ非裁量的に定量解析する新しい方法を開発した(ニュースリリース)。
国立遺伝学研究所(NIG)は,蛍光顕微鏡画像を簡便かつ非裁量的に定量解析する新しい方法を開発した(ニュースリリース)。
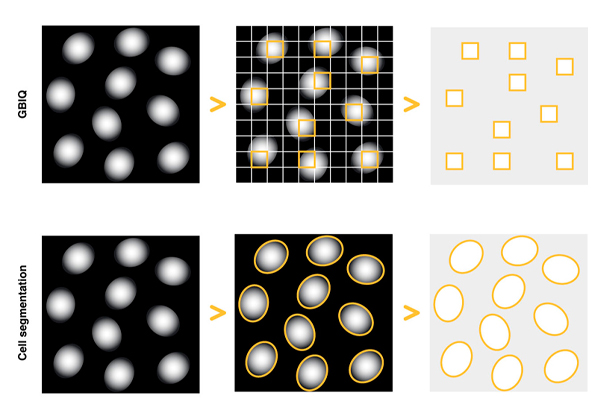
従来,細胞や組織の蛍光顕微鏡画像を定量解析するには,まず,画像内の細胞を細胞核や細胞膜などの染色像を利用して一つ一つ同定する処理cell segmentationが必要だった。この,同定された個々の細胞の蛍光強度を計測し,定量解析を行なう。
従って,ノイズやゴミなどの人為産物を取り除き,cell segmentationで適切に個々の細胞を同定することが重要となり,cell segmentationのアルゴリズムやパラメータセットを最適化する必要がある。
これは,画像定量化の最初の過程で多くのパラメータを裁量的に調整しなくてはならず,同じ画像を定量化しても,術者の選ぶパラメータセットやアルゴリズムが変わると異なった解析結果が得られることを意味する。また,現在のcell segmentationの各アルゴリズムには汎用性がなく,個々の解析対象に応じてアルゴリズムとパラメータセットを選択・最適化する必要があった。
そこで研究グループは,画像を規則的な碁盤の目状のグリッドで機械的に仕切り,各グリッドの蛍光強度の基本統計量でグリッドの蛍光特性を代表させる,簡便なアルゴリズムGBIQ(Grid Based Image Quantification)を開発した。ただし,このままでは,一つのグリッドに核と細胞質の両方が含まれるような解析には不適切なグリッドが混在するため,例えば核のみが含まれるグリッドを選択する何らかの方法が必要。
そのため,混合正規分布を個々の正規分布に分離する統計的クラス分類の手法を適用し,特定の要件を満たすグリッドのみを精度良く選択する方法(Median IQR filter)を実装した。その結果,一つのパラメータ(グリッドの大きさ)を設定するだけで,画像の定量化と解析を非裁量的に実行することに成功した。
このことは,どのような蛍光顕微鏡画像でも,GBIQを適用してパラメータを一つ決めれば,術者が誰であるかに関わらず同じ画像からは常に同じ結果が得られることを意味するという。
研究グループでは今後,GBIQをハイスループット・スクリーニングなどの大量の顕微鏡画像データの処理に応用することで,表現型の非裁量的な定量化を実現し,データ中心的な生命科学の進展に大きく寄与したいとしている。
関連記事「東工大ら,ファイバレーザを用いた二光子励起蛍光顕微鏡を実現する色素を開発」「慶大ら,SHG専用色素の開発と応用に成功」「理研ら,分子混雑が計測できる蛍光タンパク質を開発」