 理化学研究所(理研)と米国スクリプス研究所,トーマスジェファーソン大学の研究グループは,遺伝暗号解読の主要なプロセスにおいて全く新しい分子メカニズムが働いていることを発見した(ニュースリリース)。
理化学研究所(理研)と米国スクリプス研究所,トーマスジェファーソン大学の研究グループは,遺伝暗号解読の主要なプロセスにおいて全く新しい分子メカニズムが働いていることを発見した(ニュースリリース)。
人体の重要な構成要素であるタンパク質は,主に20種類のアミノ酸が連なっている。このタンパク質の合成過程では,遺伝子の塩基配列が,トランスファーRNA(tRNA)の仲介により,遺伝暗号の規則に従い特定のアミノ酸へと翻訳される。
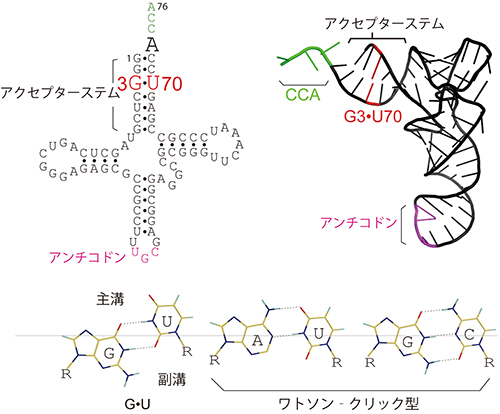
この遺伝暗号の規則を実現しているのは,個々のアミノ酸に対応する20種の「アミノアシルtRNA合成酵素(aaRS)」。aaRSは対応するアミノ酸と,そのアミノ酸の運搬を担うtRNAとを厳しく選択して結合し,反応させる。1988年に,アミノ酸の1つのアラニンで,tRNA選択の決め手(決定因子)がtRNAの二重らせん部分にある変則的塩基対(G・U塩基対)であることが発見された。しかし,アラニンに対応するaaRS「アラニルtRNA合成酵素(AlaRS)」によるtRNA選択のメカニズムには多くの謎があり,その実態は解明されていなかった。
共同研究グループは,大型放射光施設「SPring-8」と放射光科学研究施設「フォトンファクトリー」を用いて,AlaRSとそのtRNAとの複合体のX線結晶構造を解析し,tRNA選択の詳細なメカニズムの解明に成功した。アラニンと反応するtRNAの末端部分が,天然型ではAlaRSの活性部位に向かって入っていくのに対し,天然型のG・U塩基対をA・U塩基対に入れ替えた変異型tRNAでは,AlaRS上の「分岐点」で向きを変え,活性部位から遠く離れ「非反応性複合体」となることが分かった。
変異型tRNAは,天然型tRNAとほぼ同じ強さでAlaRSと結合するのにもかかわらず,反応速度が2桁も遅く,事実上選択されない。この反応速度の差だけに基づくtRNA選択の原理は,これまで説明できなかった。今回,結晶構造の解析に成功したことにより,天然型tRNAでは、G・U塩基対に起因するわずかな変形が,末端部分の位置をずらして分岐点を超えさせ,反応を2桁も速くすることが分かった。非反応性複合体を介した基質選択は新規の基本的概念となる。
研究グループは今後,これを応用することで,人工アミノ酸を遺伝暗号に組み込むなど,新技術の開発につながると期待できるとしている。