国立遺伝学研究所(NIG)は,イデユコゴメ類のC. merolaeにおいて,新たにラパマイシン誘導タンパク質ノックダウン法を開発した(ニュースリリース)。
光合成真核生物の進化の初期に分岐したグループである,単細胞紅藻イデユコゴメ類は,光合成真核生物の中で遺伝子数が最小クラスであり,細胞内構造も単純であるため,様々なオミクス解析を効率よく行なうことができる。
研究グループは,これまでにこの藻類の培養技術,相同組換えによる遺伝子破壊・導入技術,導入遺伝子の発現誘導系などを開発し,現在,世界各国の研究者等が,光合成,代謝,細胞周期,エピジェネティクス,生活環,タンパク質の構造,進化,産業利用など多様な分野の研究で,この藻類を用い始めている。
一方,イデユコゴメ綱の藻類は,RNA干渉機構(RNAi)を担う遺伝子群を失っているため,その機構を利用した遺伝子ノックダウンの実験を行なえず,増殖や生存に必須な遺伝子の機能解析が困難だった。
これまで酵母や動物細胞を用いた研究では,RNA干渉に代わる手法として,目的遺伝子を狙ったタイミングで分解させる,誘導型タンパク質ノックダウン法が開発されてきた。
この手法は,新規のタンパク質合成を阻害するRNAiとは異なり,すでに合成済みで細胞内で機能している標的タンパク質を直接分解できるという利点を有している。しかしながら,植物ホルモンであるオーキシンは植物の成長や形態形成に影響を与え,またイデユコゴメ類に対して毒性があるため,この手法は適用できなかった。
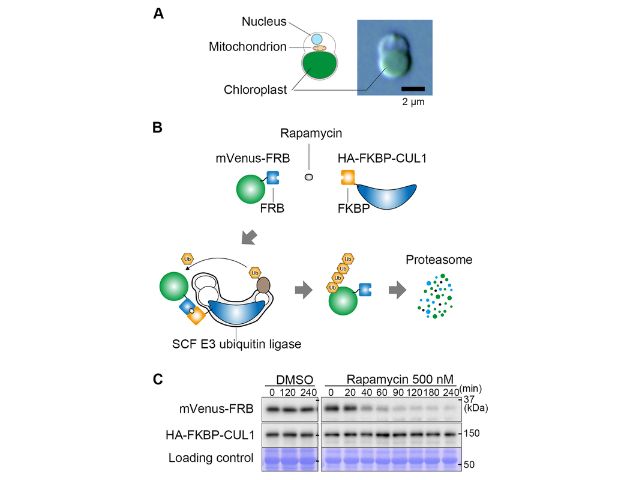
そこで研究グループは,イデユコゴメ類のC. merolaeにおいて,新たにラパマイシン誘導タンパク質ノックダウン法を開発した。この手法は,化学物質ラパマイシンが,FRBタグ(ペプチド)とFKBPタンパク質を結合させる性質を利用している。
標的タンパク質にFRBを,ユビキチンリガーゼのサブユニット(CUL1)にFKBPを融合した細胞を用意し,これにラパマイシンを与えると,標的タンパク質とユビキチンリガーゼ複合体が結合し,標的タンパク質のユビキチン化とその後の分解が起こる。
この手法を用いると,例えば試験用の標的タンパク質はラパマイシン添加後,2時間以内にほとんど分解される。また,内在の必須遺伝子であるDRP5B(葉緑体分裂タンパク質)や核内の細胞周期制御因子E2Fを迅速に分解できることを示した。
研究グループは,この方法は藻類や陸上植物を含む光合成真核生物における精緻な実験系の構築を可能にし,様々な生物現象の解明に貢献するとしている。