 岡山大学大学の研究グループは,サツマイモの“動く遺伝子”に注目し, “次世代シーケンス”とよばれる技術を組み合わせることで効率的な遺伝解析を行なった(ニュースリリース)。これにより,サツマイモ品種間の遺伝関係を明らかにし,また品種を特定するためのDNAマーカーの開発に成功した。
岡山大学大学の研究グループは,サツマイモの“動く遺伝子”に注目し, “次世代シーケンス”とよばれる技術を組み合わせることで効率的な遺伝解析を行なった(ニュースリリース)。これにより,サツマイモ品種間の遺伝関係を明らかにし,また品種を特定するためのDNAマーカーの開発に成功した。
サツマイモは,他の農作物と比べて遺伝解析が非常に難しく,DNAマーカーの開発が遅れていた。その理由としては,サツマイモが倍数体(六倍体)であり,遺伝が複雑なため。さらに,その全ゲノム配列情報もいまだに解読されていない,などが挙げられる。しかし,有用な品種を効率的に育成し,育成者の特許権を守るためには,遺伝情報を用いた解析手法の確立が必要不可欠だった。
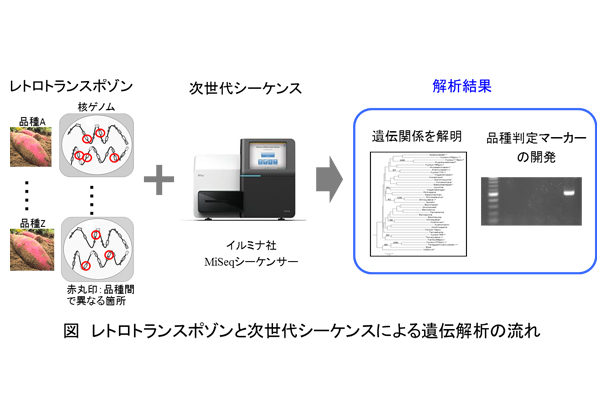
そこで今回,研究グループは,レトロトランスポゾンとよばれる“動く遺伝子”に注目した。これは,真核生物(植物・動物・菌類など)のゲノム中に存在し,自分のコピーを作って増えることができる。また,新しく作られたコピーは,一度DNA配列中に入ると安定して遺伝する。そのため,品種間で異なる場所にあるコピーは,DNAマーカーとして使うことができる。
また,研究グループは効率的な遺伝解析を行なうため,近年急速に普及した“次世代シーケンス”を利用。短期間で数千万という非常に多くのDNA配列のデータを得ることができた。そして,解析結果からサツマイモ品種間の遺伝的な関係を明らかにし,すでに知られていた品種間の親子関係とほぼ一致することを確認。今まで困難であったサツマイモにおいて,得られた遺伝子データから品種管理ができることを実証した。さらに,品種を特定するためのDNAマーカーの開発にも成功した。これにより,より的確な品種の特定を行なうことが出来るようになった。
関連記事「京大ら,コムギのゲノム配列の概要解読に成功」「NIAS,イネの高温耐性に関わる遺伝領域を解明しDNAマーカーを開発」